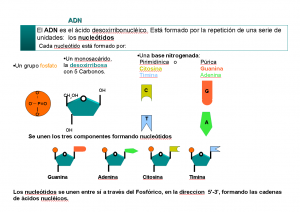
Hoy, 25 de abril se conmemora el descubrimiento de la estructura del ADN por Watson y Crick, basado también en el trabajo de Rosalind Franklin, de la que el día 16 hizo 57 años de su muerte.
Algunas curiosidades:
- Si multiplicamos la longitud del DNA de una célula por todas las células del ser humano, haríamos 70 viajes de ida/vuelta al sol.
- ¿Sabías que el ADN te influye a la hora de escoger pareja?
- El ADN contiene más información en un centímetro cúbico que un millón de millones de Compact Discs
- Sí “estirásemos” el ADN de cada célula alcanzaríamos una longitud de 2 metros
- Si estirásemos el ADN de todas las células de nuestro cuerpo, con su longitud ¡podríamos dar más de 1.500 vueltas al planeta!
- 1 millon de bases(Megabases) de DNA es el equivalente a 1 Megabyte de almacenamiento en un pc
- El ADN mitocondrial sólo se hereda de la madre.
- Si nuestro genoma fuese un libro y se leyesen 10 letras por segundo ¡tardaríamos 11 años en leerlo!
- Sabías que si pusiésemos todo el ADN de un ser humano en línea recta llegaría a Plutón.
- El helecho indio tiene mas de 1000 pares de cromosomas mientras que el ser humano solo 23
- ¿Sabías que para escribir todas las letras que forman el genoma humano harían falta más de 214 millones de tuits?
- ¿Sabías que para escribir todas las letras que forman el genoma humano harían falta más de 214 millones de tuits?
- Si multiplicamos la longitud del DNA de una célula por todas las células del ser humano, haríamos 70 viajes de ida/vuelta al sol.
¿Qué podemos hacer con los alumnos?
1. Al igual que se hace en el día del libro con El Quijote, se puede leer un capítulo del libro de Genoma, Matt Ridley. Es un libro que tiene 23 capítulos, uno por cada cromosoma y que elige un gen controvertido para discutir sobre lo que es genético o no. Das con ello pie a fomentar la lectura divulgativa, que no es poco, y a la discusión: hasta donde llega la Ciencia, lo que hoy es verdad, mañana no lo es…
2. Recorta y pega del ADN: hay varios modelos.
http://cienciasnaturalesgtb.wikispaces.com/file/view/adn+1.pdf
http://es.scribd.com/doc/67760648/ADN-RECORTABLE
3. Lectura del texto original del descubrimiento: http://www.bioxeo.com/adn.htm
4. Trabaja con la línea de tiempo sobre la Historia del ADN: tarda bastante en bajar:
http://www.biologia.edu.ar/macromoleculas/macromedia/history.exe
5. Juega con el ADN.
Puedes construir una molécula http://learn.genetics.utah.edu/es/units/basics/builddna/
Traduce y transcribe un gen: http://learn.genetics.utah.edu/es/units/basics/transcribe/posteriormente, un gen.
6. Proyecto Genoma humano: http://www.genome.gov/25019879. Aquí te explican tambiéncómo se secuencia el ganoma: http://www.genome.gov/Edkit/flash/intro.html Tiene un montón de animaciones.
7. Puedes extraer ADN: http://learn.genetics.utah.edu/es/units/activities/extraction/
8. Revisa las animaciones sobre el ADN: http://www.elmundo.es/especiales/2003/02/salud/genetica/descifrar_la_vida.html
1. Lectura de “Hasta ahora se creía que las únicas moléculas capaces de contener y transferir información biológica eran el ADN y el ARN. Un equipo de científicos ha sintetizado en el laboratorio seis polímeros que también cumplen con las leyes de la herencia y, uno de ellos, con la evolución darwiniana.” Sigue leyendo
2. Dibuja, fabrica, fotografía ADN:
3. Visualiza este video: https://www.youtube.com/watch?v=VZ8GZRx5_Vk

«ADN animation» por brian0918™ – Trabajo propio. Disponible bajo la licencia Dominio público vía Wikimedia Commons.